GODDESS / GRASS
Разработанные нами программы для симулирования спектров ЯМР олиго- и полисахаридов и родственных соединений и предсказания структуры по данным ЯМР подразумевают два аспекта: предсказание спектров по структурам и предсказание структур по спектрам.Предсказание спектров ЯМР 13C и 1H углеводов
Glycan-Optimized Database-Driven Empirical Spectra Simulation (GODDESS, "Основанное на базе данных эмпирическое моделирование спектров, оптимизированное для углеводов") объединяет эмпирические подходы к предсказанию спектров ЯМР 13C:1. Инкрементная схема (только 13C) предсказыввает подспектры мономерных остатков, используя специальные спектральные базы данных по моно-, ди- и тримерным фрагментам, теоретическим эффектам замещения и стерическим поправкам. Изначально она была разработана в рамках проекта Biopolymer Structure Elucidation (BIOPSEL) в 2001-м году. С тех пор она была усовершенствована, адаптирована к большему разнообразию структур (в том числе включающих неуглеводные компоненты) и приобрела веб-интерфейс. В 2013-м году мы показали, что этот алгоритм значительно превосходит используемые в ЯМР-исследованиях квантовомеханические подходы (напр., geometry+giao: B3LYP/6-311G++(2d,2p) и PBE/PBE) как по точности предсказания химических сдвигов 13С углеводов в водных растворах, так и по быстродействию [ref]. Нажмите здесь для более подробной справки на сайте проекта.
2. Статистическая схема (13C и 1H), адаптирующая подход HOSE к атомным группировкам и использующая эвристический алгоритм генерализации структуры, оптимизированный для углеводов. Этот подход не требует специальных баз данных, а использует "большую" регулярно пополняемую базу CSDB (>4000 спектров). Алгоритм обощает структурное окружение предсказываемого атома до тех пор, пока в базе не будет найдено достаточно структурно-близких фрагментов, и усредняет химические сдвиги с учетом выбросов. В зависимости от характера обобщения, его стереохимии и удаленности от предсказываемого атома, ему придается определенный вес. Критерием нахождения оптимального набора обощений является их минимальный общий вес. В 2015 средняя точность предсказания на природных углеводах и гликоконъюгатах составила 0.86 м.д. для 13C и 0.07 м.д. для 1H [ref]. Нажмите здесь для более подробной справки на сайте проекта.
3. Как инкрементная, так и статистическая схема оценивают достоверность и точность каждого конкретного предсказания. На основании этих значений, полноты выборки и разброса данных в базе, гибридная схема (только 13C) комбинирует результаты двух подходов, используя гибкие коэффициенты. Нажмите здесь для более подробной справки на сайте проекта.
Статистическая и гибридная схемы позволяют проследить использованные данные и обобщения вплоть до исходных публикаций. Оба подхода реализованы в виде модулей Базы данных углеводных структур (CSDB). Чтобы ввести структуру и запустить симуляцию, нажмите Extras/Predict NMR в меню CSDB или используйте прямую ссылку: NMR simulation. Сервис генерирует одно- и двумерные спектры ЯМР и таблицы отнесения сигналов, примерно так:
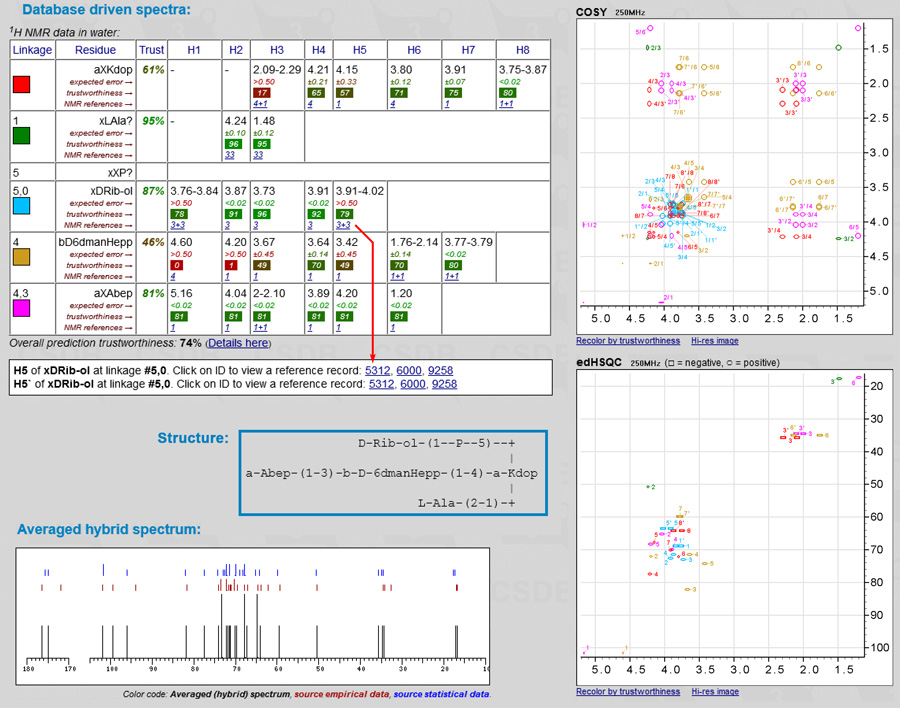
В настоящее время поддерживается схематическая визуализация спектров 1D 13C, COSY, COSY RCT, COSY DQF, TOCSY, edHSQC, HSQC-TOCSY и HMBC для произвольной рабочей частоты. Точность предсказания на двух типичных структурах отражена на рисунках:
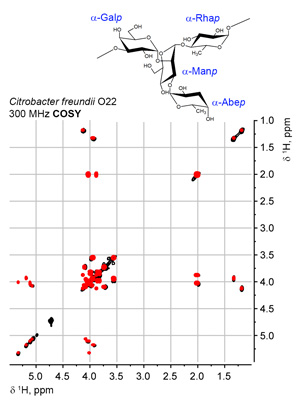
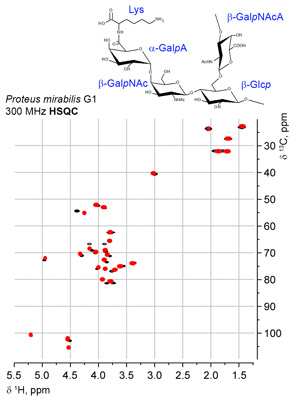
Здесь можно посмотреть презентацию сервиса GODDESS на Балтийской Встрече по Углеводам Микробов в Ростоке, 2016 (PDF, слайды и доклад).
Предсказание углеводных структур по спектрам ЯМР
Generation, Ranking and Assignment of Saccharide Structures (GRASS, "генерирование, ранжирование и отнесение структур сахаридов") - это итератор структур, перебирающий все возможные варианты строения олигосахарида, полисахарида или гликоконъюгата в пределах заданных структурных ограничений. Обязательными параметрами являются неотнескенный экспериментальный спектр ЯМР 13C и число остатков в олигомере или повторяющемся звене полимера. Точность предсказания может быть улучшена указанием дополнительных данных, таких как число CH2-групп, общее число β-сахаров, данные метилирования, мономерный состав (GC), абсолютные, аномерные конфигурации и размеры циклов и т.д. Быстрый эмпирический симулятор спектров ЯМР 13C вызывается для каждой генерируемой структуры, после чего спектры ≤500 наилучших структурных гипогтез уточняются медленным, но более точным статистическим методом. Алгоритм толерантен к неточной оцифровке спектра ЯМР (лишние или пропущенные сигналы). Структурные гипотезы ранжируются по степени сходства предсказанного и экспериментального спектров:
| ВВОД: |
ПРЕДСКАЗАНИЕ:| ВЫВОД: |
|
